・例えば、化合物の構造だけ与えられて、「これと似たような化合物が、何かの生理活性を測定された報告はあるのかな?」と気になったとする。
・個別の化合物を検索するのであれば、CHEMBL(以下)などでSmilarityも含めて、アッセイ結果を検索してみるのが良い。
・化合物が50個や100個ある場合は、個別に検索するのはハードなので、自動化したいところ。
・ChemblのAPI(以下)と、KNIMEのGet Request、Xpathなどを使えばなんとかなりそうな気がする。
・流れとしては、①SMILESからChembl_IDを釣ってくる、②Chembl_IDから評価情報を釣ってくる、という感じにしたい。
・まずは①を組んでみた(下図)。
・SMILS情報は、以下の通り。
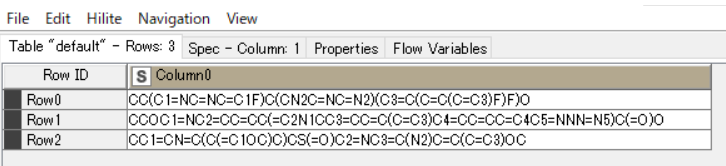
・SMILESの表記方法として、Canonical SMILESを採用している。一番上の化合物だと、Canonical SMILESでCC(C1=NC=NC=C1F)C(CN2C=NC=N2)(C3=C(C=C(C=C3)F)F)Oと表記され、Isomeric SMILESでC[C@@H](C1=NC=NC=C1F)[C@](CN2C=NC=N2)(C3=C(C=C(C=C3)F)F)Oと表記される。Get Requestノードでは、"["や"]"がエラーになるので、Isomeric SMILESだとうまくワークしない。
・String Manipulationは以下の通り。
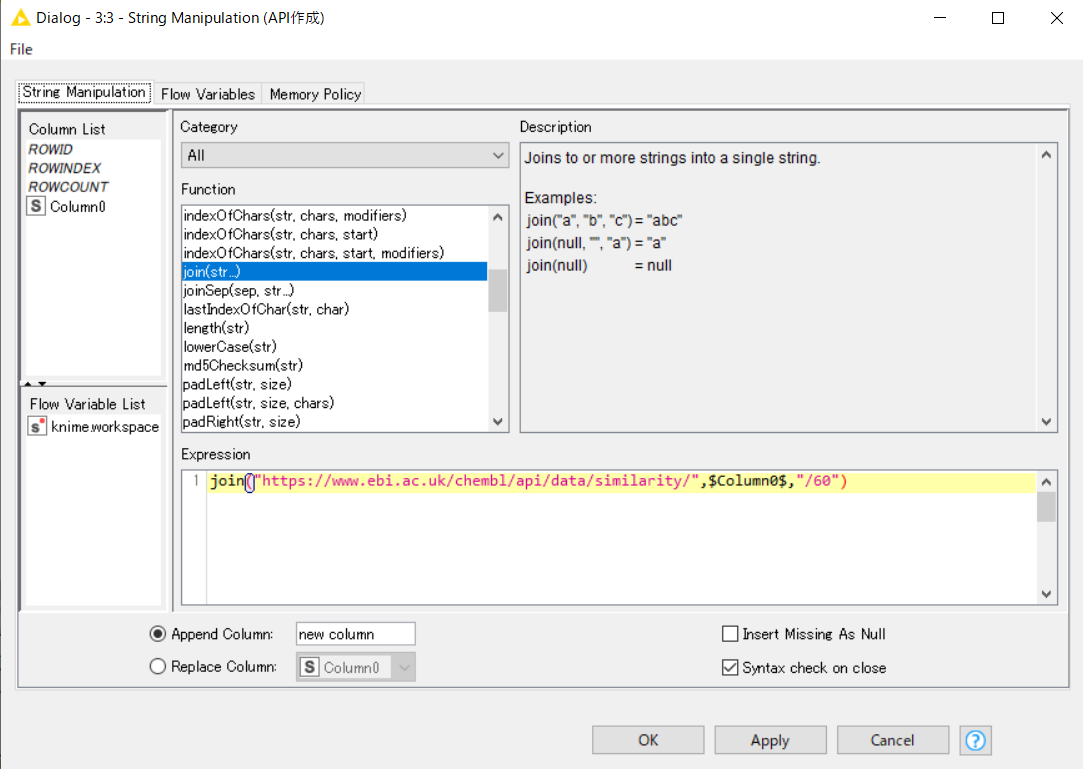
・"/60"はSimilarityを表しており、"/100"にすると完全に一致している場合しかヒットしなくなる。
・Get Requestは以下の通り。
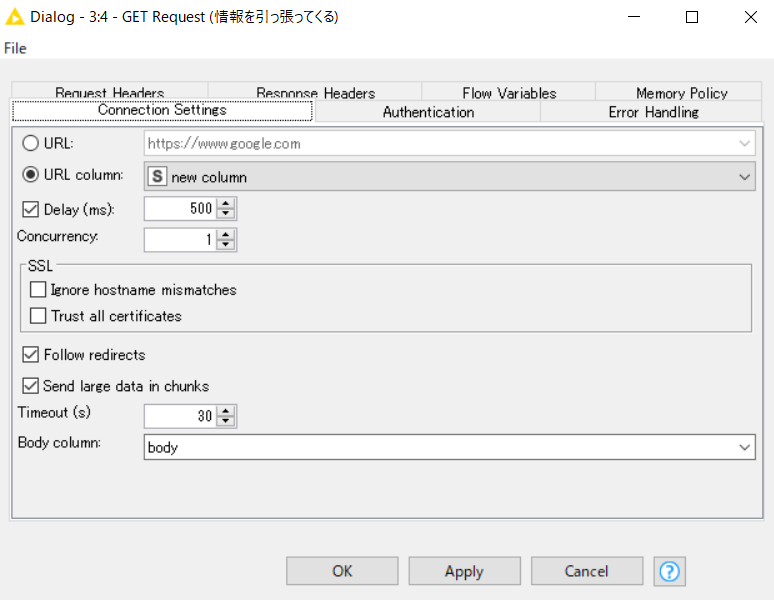
・Delayは十分に長くとっておかないと、サーバーに負荷をかけるので、500msに設定しておいた。Timeoutは、ある程度長めに取っておく必要がある。30秒程度がおすすめ。
・次に、Xpathでchembl_idを抜き出した(下図)。
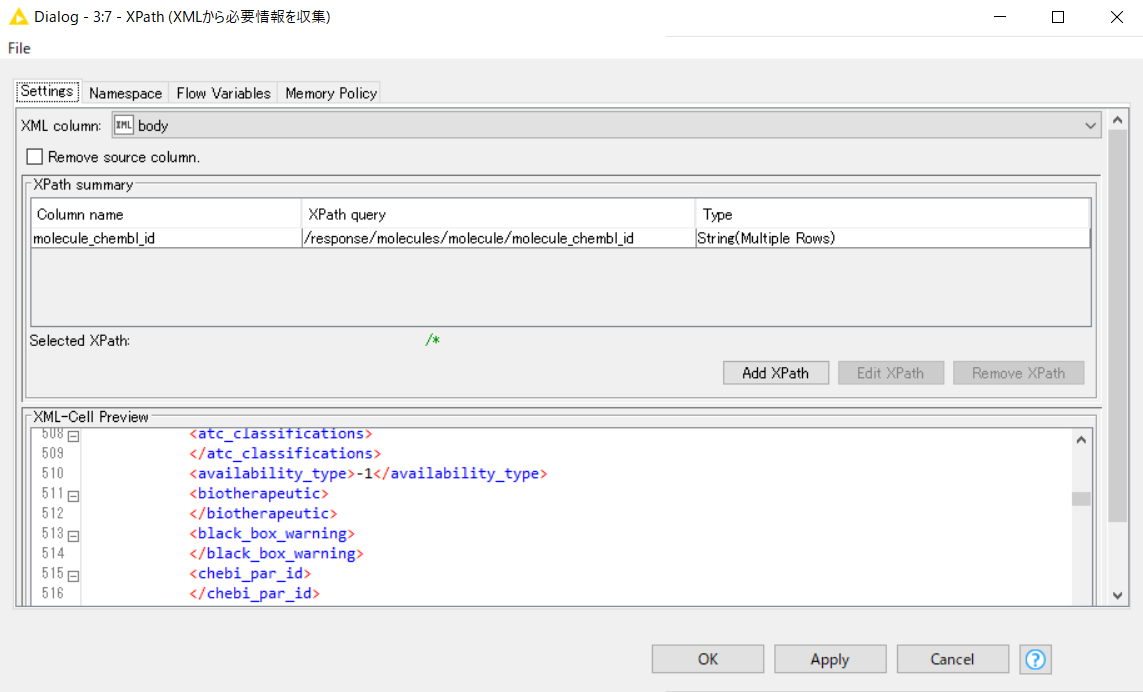
・Multiple tag optionsはMultiple Rowsにする。
・Xpathの出力は以下の通り。目的のものらしいchembl_idが得られた。

いったん終わり。