・お題:元ベクトルに対応するように色を振ったベクトルを作りたい。
・やることは、元ベクトルをファクター化して、色のベクトルから要素をとって来るような感じ。
> ori <- c("1","1","2","3","3") #元ベクトルを設定
> iro <- c("red", "blue", "yellow") #色ベクトルを作成
> iro[factor(ori)]
[1] "red" "red" "blue" "yellow" "yellow"
・試しにヒートマップを作るのに使ってみる。
> library(tidyverse) #便利に使うライブラリ
> library(gplots) #heatmap.2のライブラリ
・適当にデータセットを作る。
> df <-
+ rbind(
+ data.frame(
+ sample_1 = rnorm(n = 10, mean = 100, sd = 20),
+ sample_2 = rnorm(n = 10, mean = 100, sd = 20),
+ sample_3 = rnorm(n = 10, mean = 100, sd = 20),
+ sample_4 = rnorm(n = 10, mean = 80, sd = 10),
+ sample_5 = rnorm(n = 10, mean = 80, sd = 10),
+ sample_6 = rnorm(n = 10, mean = 120, sd = 30),
+ sample_7 = rnorm(n = 10, mean = 120, sd = 30),
+ row.names = paste0("gene", LETTERS[1:10])
+ ),
+ data.frame(
+ sample_1 = rnorm(n = 10, mean = 70, sd = 10),
+ sample_2 = rnorm(n = 10, mean = 70, sd = 10),
+ sample_3 = rnorm(n = 10, mean = 70, sd = 10),
+ sample_4 = rnorm(n = 10, mean = 130, sd = 30),
+ sample_5 = rnorm(n = 10, mean = 130, sd = 30),
+ sample_6 = rnorm(n = 10, mean = 100, sd = 20),
+ sample_7 = rnorm(n = 10, mean = 100, sd = 20),
+ row.names = paste0("gene", LETTERS[11:20])
+ )
+ )
> df %>% head(.)

・サンプル構成を作る。
> group <-
+ rep(c("alpha","beta","gamma"), c(3,2,2)) %>%
+ factor(.)
> group
[1] alpha alpha alpha beta beta gamma gamma
Levels: alpha beta gamma
・カラーセットを作る。色は以下を参考にした。
> colorBlindGrey8 <-
+ c("#999999", "#E69F00", "#56B4E9", "#009E73",
+ "#F0E442", "#0072B2", "#D55E00", "#CC79A7")
・それぞれのサンプルに対応する色のベクトルを作る。
> colors <- colorBlindGrey8[group]
> colors
[1] "#999999" "#999999" "#999999" "#E69F00" "#E69F00" "#56B4E9" "#56B4E9"
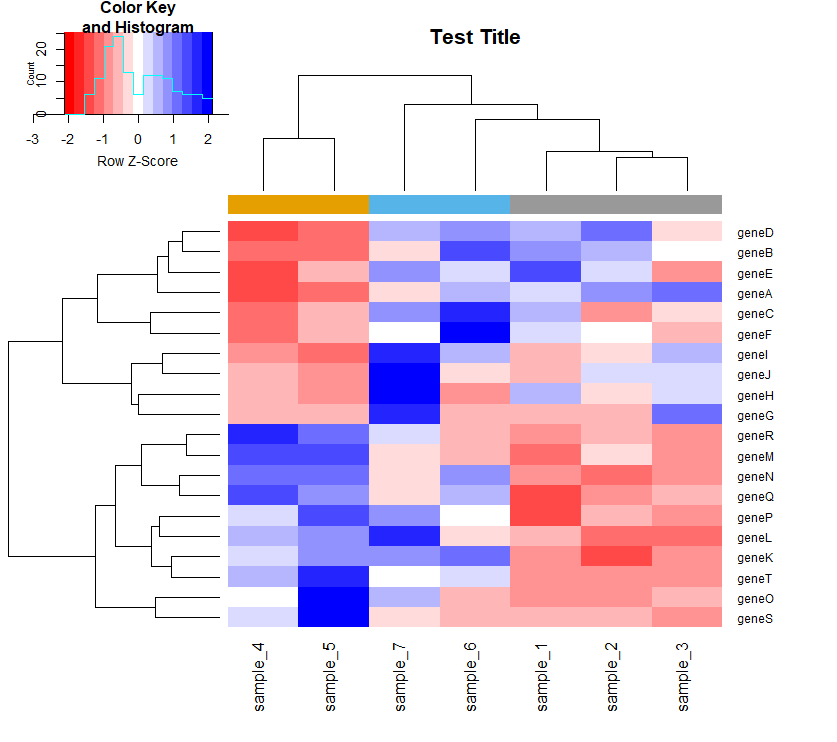
・ヒートマップを描いてみる。引数marginsに余白を指定しないとsampleが見切れてしまう。
> heatmap.2(x = as.matrix(df),
+ trace = "none",
+ main = "Test Title",
+ ColSideColors = colors,
+ col = "redblue",
+ scale = "row",
+ margins = c(8,8))
・グラデーションを作るなら、gplotsパッケージのcolorpanel関数を使うのも有効。
> library(gplots)
> col <- colorpanel(1000,"blue","white","red") #色を指定しておく。
おわり