・お題:バイオインフォマティクスなどのデータを可視化するOSSプラットフォームで、Cytoscapeというやつがあるらしい。Rでこれを弄ってみたい。
・今回は、前回の続きとして、以下のチュートリアルのVisualizing data on networks以降の内容を進めていく。正しいことは元サイトをご確認いただきたい。
・ライブラリを読み込む。
> library(RCy3)#これがCytoscapeを操作するライブラリ
> library(tidyverse)
> library(RColorBrewer)
>installApp('STRINGapp')
・データをロードする。今回はBreast Cancerのデータセット。
> load(system.file("extdata","tutorial-brc-expr-mean-dataset.robj", package="RCy3"))
> load(system.file("extdata","tutorial-brc-mut-dataset.robj", package="RCy3"))
・データセットの中身を見てみる。
> str(brc.expr) # 行名の遺伝子名が入っている発現量のデータフレーム
'data.frame': 17508 obs. of 1 variable:
$ expr.mean: num -0.000167 -0.005408 0.018381 -0.065037 -0.074444 ...
> str(brc.mut) # Hugo_Symbolに遺伝子名が入っている変異のデータフレーム
'data.frame': 12463 obs. of 3 variables:
$ Hugo_Symbol : Factor w/ 14130 levels "1-Dec","1-Sep",..: 16 17 18 19 20 22 23 24 25 26 ...
$ mut_count : int 2 3 3 4 1 2 1 3 2 1 ...
$ missense_mut_count: num 2 3 2 4 0 2 1 2 1 1 ...
・データのロードに関するルールを設定しておく。
> loadTableData(brc.expr, table.key.column = "display name") #default data.frame key is row.names
[1] "Success: Data loaded in defaultnode table"
> loadTableData(brc.mut, 'Hugo_Symbol', table.key.column = "display name") #specify column name if not default
[1] "Success: Data loaded in defaultnode table"
・今回は、この情報をSTRINGの相互作用ネットワークにマッピングして可視化することになる。
・STRINGのbreast cancer関連の相互作用ネットワークを読み込む。
>string.cmd = 'string disease query disease = "breast cancer" cutoff = 0.9 species = "Homo sapiens" limit = 150'
>commandsRun(string.cmd)
> setCurrentNetwork(network = "STRING network - breast cancer")
> layoutNetwork('force-directed')

・STRINGっぽいスタイルのネットワークになっている。スタイルを変更してみる。
> style.name = "dataStyle"
> createVisualStyle(style.name)
> setVisualStyle(style.name)
message
"Visual Style applied."

・さらに細かい表示設定を弄っていく。
> setNodeShapeDefault("ellipse", style.name) #remember to specify your style.name!
> setNodeSizeDefault(60, style.name)
> setNodeColorDefault("#AAAAAA", style.name)
> setEdgeLineWidthDefault(2, style.name)
> setNodeLabelMapping('display name', style.name)
NULL
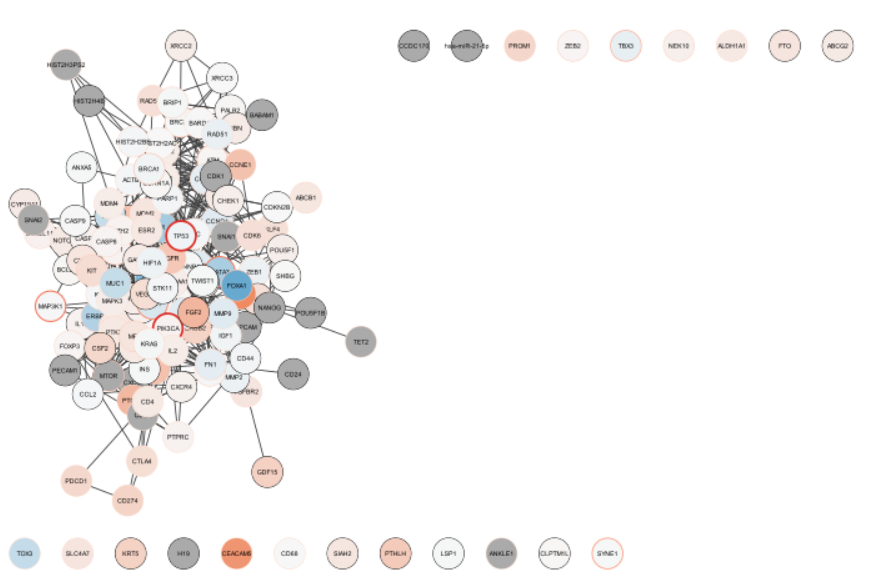
・次に、遺伝子発現量に応じて色を変えていく。遺伝子発現量の情報は、brc.exprにexpr.meanとして格納されている。
> brc.expr %>% head()

・データを読み込む。
> brc.expr.network = getTableColumns('node','expr.mean')
・配色を設定するために、発現量の最大値と最小値の情報を抽出する。
> min.brc.expr = min(brc.expr.network[,1],na.rm=TRUE)
> max.brc.expr = max(brc.expr.network[,1],na.rm=TRUE)
> data.values = c(min.brc.expr,0,max.brc.expr)
・配色を設定する。
> display.brewer.all(length(data.values), colorblindFriendly=TRUE, type="div") # div,qual,seq,all
> node.colors <- c(rev(brewer.pal(length(data.values), "RdBu")))
・先の配色でネットワークを塗る。
> setNodeColorMapping('expr.mean', data.values, node.colors, style.name=style.name)
NULL

・変異の情報に関しても、同様に図示する(枠を塗る)。
> brc.mut.network = getTableColumns('node','mut_count')
> min.brc.mut = min(brc.mut.network[,1],na.rm=TRUE)
> max.brc.mut = max(brc.mut.network[,1],na.rm=TRUE)
> data.values = c(min.brc.mut,20,max.brc.mut)
> display.brewer.all(length(data.values), colorblindFriendly=TRUE, type="seq")
> border.colors <- c(brewer.pal(3, "Reds"))
> setNodeBorderColorMapping( 'mut_count', data.values, border.colors, style.name=style.name)
NULL
> border.width <- c(2,4,8)
> setNodeBorderWidthMapping( 'mut_count', data.values, border.width, style.name=style.name )
NULL
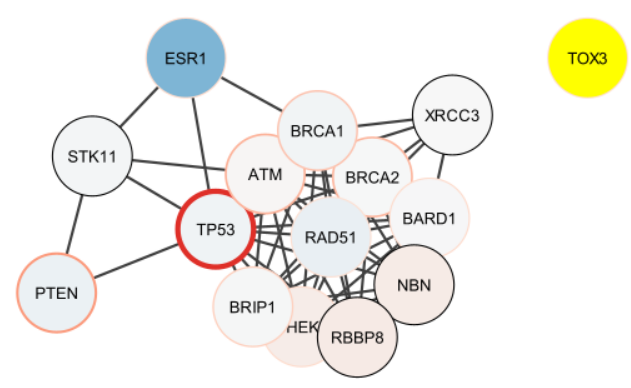
・あまりに多すぎるので、disease scoreトップ3から始まる拡散アルゴリズムで、ネットワークの接続性を出してみる。
> setCurrentNetwork(network="STRING network - breast cancer")
> disease.score.table <- getTableColumns('node','stringdb::disease score')
> disease.score.sorted <- disease.score.table[order( -disease.score.table$`stringdb::disease score`), , drop = FALSE]
> top.nodes <- row.names(disease.score.table)[tail(order(disease.score.table[,1]),3)]
> selectNodes(nodes = top.nodes)
$nodes
[1] 2039 2024 2030
$edges
list()
> commandsPOST('diffusion diffuse') # diffusion!
heatColumn rankColumn
"diffusion_output_4_heat" "diffusion_output_4_rank"
> createSubnetwork('selected',subnetwork.name = 'top disease diffusion')
network
17453
> layoutNetwork('force-directed')
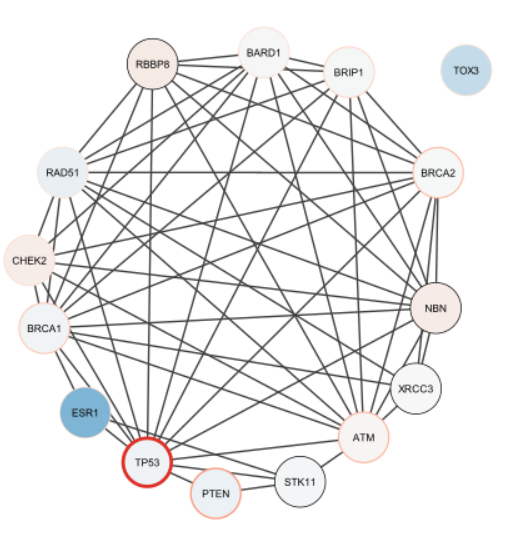
> layoutNetwork('circular')
・ちなみに、出てきたネットワークはインタラクティブなものとして保存できる。RCy3でのコマンドが良く分からなかったので、Cytoscapeからexportした。

・これで、zipファイルが作成される。ネットワークを開くときは、zipを解凍し、index.htmlを適当なブラウザで開く。インタラクティブなので、ノードを動かすことができる。
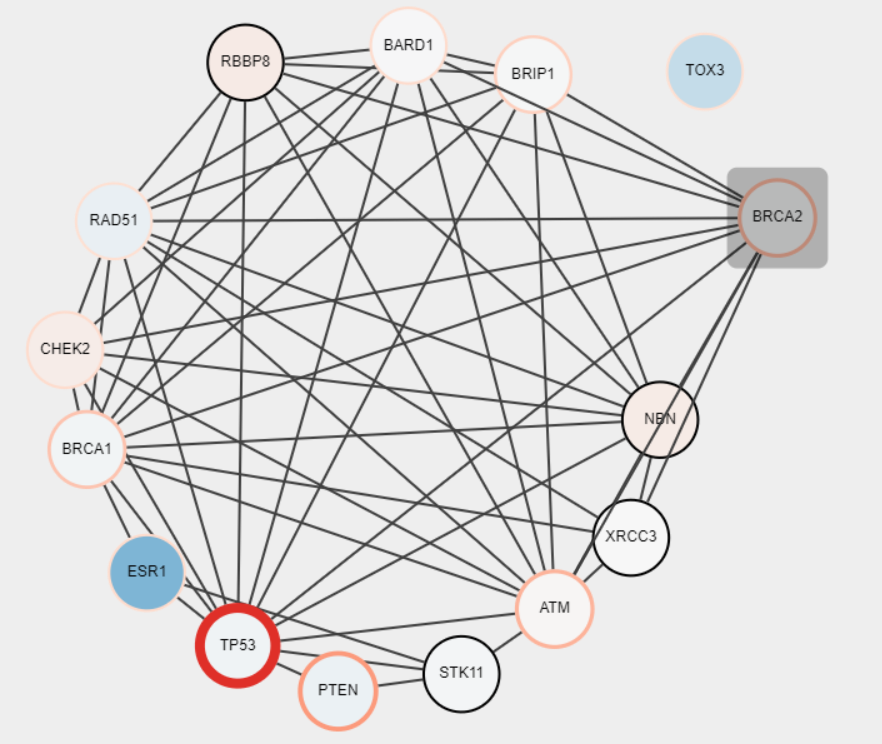
つづく。