・お題:バイオインフォマティクスなどのデータを可視化するOSSプラットフォームで、Cytoscapeというやつがあるらしい。Rでこれを弄ってみたい。
・チュートリアルを探してみたところ、以下のサイトが分かりやすそうだったので、いつものようになぞって勉強させていただく。正しいことは元サイトをご確認いただきたい。
・Cytoscapeのソフトウェアとライブラリを起動する。
> library(RCy3)
> library(tidyverse)
> library(RColorBrewer)
> installApp('STRINGapp')
App STRINGapp installed
> installApp('enhancedGraphics')
App enhancedGraphics installed
> installApp('clusterMaker2')
App clusterMaker2 installed
・今回は、Exercise 3からやってみる。
・データは以下の報告で見出された上皮性卵巣癌 (EOC) に関連する541のタンパク質のリストを用いる。
・データを読み込む。
> eoc.df <- read.table("https://raw.githubusercontent.com/cytoscape/cytoscape-automation/master/for-scripters/R/notebooks/stringApp/Francavilla2017CellRep.tsv",
+ header = TRUE,
+ sep = "\t",
+ quote="\"",
+ stringsAsFactors = FALSE,
+ check.names = FALSE)
> eoc.df %>% head()

・遺伝子名などの情報が入っている。遺伝子名はUniProt列に入っている。STRINGでこれらの遺伝子の相互作用を釣ってきて、ネットワークを作成する。
・STRINGからこれらの遺伝子の情報を釣ってくるために、クエリを作成する。
> string.cmd = paste('string protein query query="', paste(eoc.df$UniProt, collapse = '\n'), '"', sep = "")
> string.cmd

・クエリを用いてデータを落としてくる。cytoscapeでなんだかカクカクしたグラフができた。
> commandsRun(string.cmd)
[1] "Loaded network 'STRING network' with 547 nodes and 5053 edges"

・カクカクしているのは解像度(?)の問題で、ズームすればまん丸のノードを見ることができる。
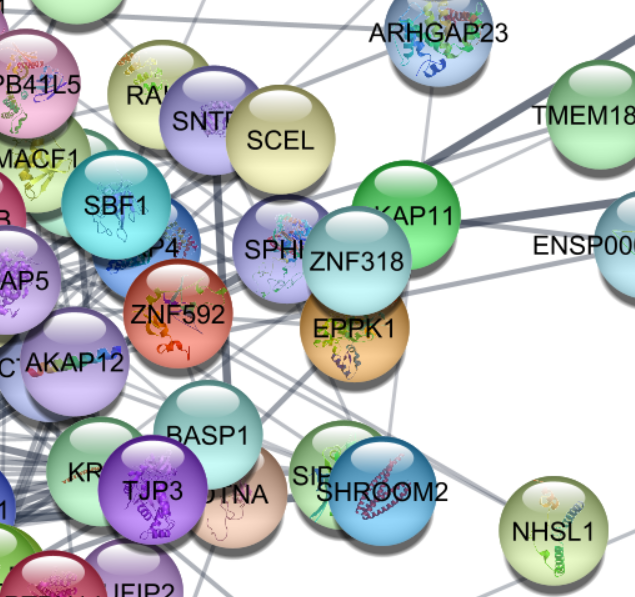
・名前を変えておく。
> renameNetwork("EOC - data")
・レイアウトを変更してみる。
> layoutNetwork('degree-circle')

> layoutNetwork('force-directed edgeAttribute="score"')#ここでいうscoreはSTRINGのネットワークスコアのことっぽい。

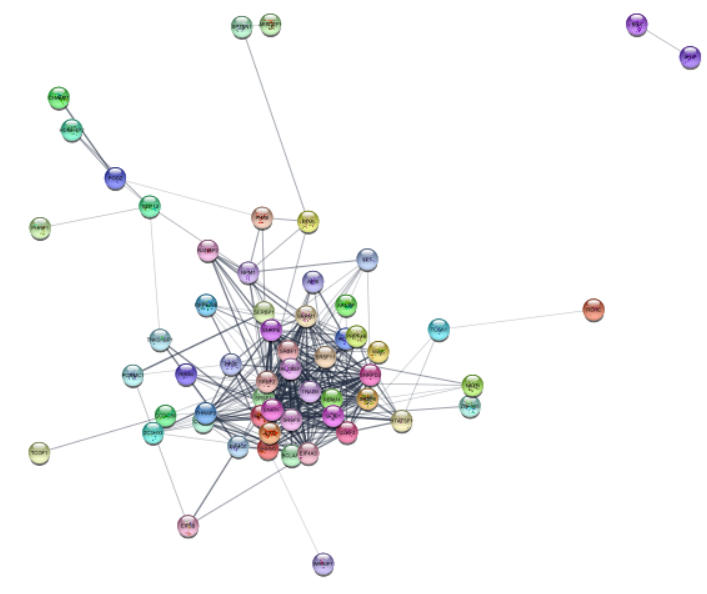
・さすがに多すぎるので、少し絞る。p<0.001のノードを元データから抽出し、それをもとにグラフを作成しなおす。ついでに、繋がっていないものを省く。
> eoc.df2 <- eoc.df %>%
+ rename(., "AdjPvalue" = "Adj p-value") %>%
+ filter(., AdjPvalue < 0.001)
> string.cmd = paste('string protein query query="', paste(eoc.df2$UniProt, collapse = '\n'), '"', sep = "")
> commandsRun(string.cmd)
[1] "Loaded network 'STRING network - 1' with 74 nodes and 338 edges"
> renameNetwork("EOC - data 2")
> createSubnetwork(edges='all',subnetwork.name='EOC - data 2_connected')
network
31253
> layoutNetwork('degree-circle')
・少し見た目を変える。
> style.name = "dataStyle"
> createVisualStyle(style.name)
> setVisualStyle(style.name)
message
"Visual Style applied."

・デフォルトが真っ赤なので、グレーにする。
> setNodeColorDefault("#AAAAAA", style.name)
・色を塗る。今回は、Epigeneticを赤色に、Kinaseを青色に塗る。
> column <- "target::family"
> values <- c('Epigenetic', 'Kinase')
> colors <- c('#FF0000', '#0000FF')
> setNodeColorMapping(column,
+ values,
+ colors,
+ mapping.type = "d",
+ style.name = style.name)
NULL

・ラベルもつける。見づらいので切り出すと以下のような感じ。
> setNodeLabelMapping('display name', style.name)
NULL

・これで、DEGの遺伝子名を使ってSTRINGから相互作用を釣ってきてネットワークを形成し、形成したネットワークにクラスをラベルすることができた。
・次に、このネットワークにSTRING外の情報を付与し、それを図示することを考える。今回付与する情報はeoc.dfを想定する。付与するデータフレーム、データフレーム側のキーの列名、ネットワーク側の表、ネットワーク側のキーの列名を指定する。
> loadTableData(as.data.frame(eoc.df), data.key.column = "UniProt", table = "node", table.key.column = "query term")
[1] "Success: Data loaded in defaultnode table"
・本当に情報が付与できたが見てみると、お尻の方に確かに情報が加わっているっぽい。
> getTableColumns('node') %>% str()
前略

・EOC.vs.FTE.OSEの数値データを使って、ネットワークに色を着けてみたい。とりあえずデータを切り出す。よく見ると、eoc.dfの列名から少し変わっている。スペースや&が受け入れられず、ドットに変換されているっぽい。
> logratio.score.table <- getTableColumns('node', "EOC.vs.FTE.OSE")
> logratio.score.table %>% head()

・EOC.vs.FTE.OSEの数値データを使ってグラデーションをつけるので、数値データを色データに変換する必要がある。最小値と最大値を求めて、ゼロを中心にカラーセット(の端っこと真ん中)を作成する。
> logratio.min <- min(logratio.score.table, na.rm = T)
> logratio.max <- max(logratio.score.table, na.rm = T)
> logratio.data.values = c(logratio.min, 0, logratio.max)
> logratio.data.values
[1] -4.30 0.00 9.25
> logratio.node.colors <- c(brewer.pal(length(logratio.data.values), "RdBu"))
> logratio.node.colors
[1] "#EF8A62" "#F7F7F7" "#67A9CF"
・色を塗る。
> getCurrentStyle()
[1] "dataStyle"
> setNodeColorMapping('EOC.vs.FTE.OSE',
+ logratio.data.values,
+ logratio.node.colors,
+ style.name = "dataStyle")
NULL
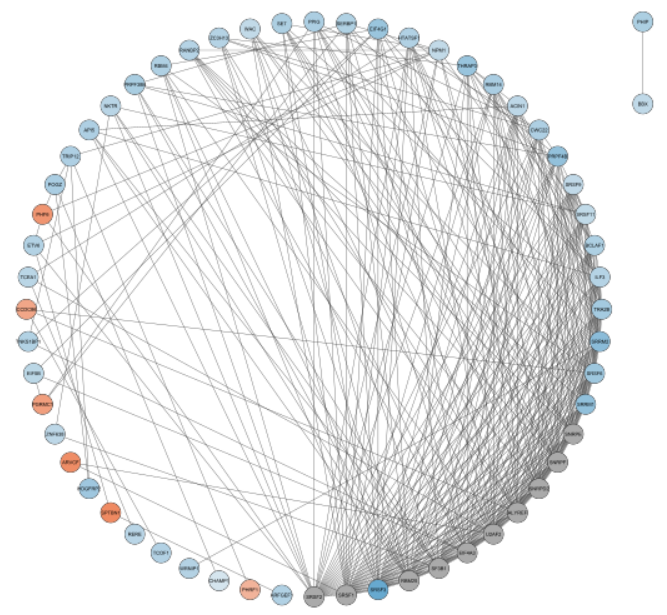
・次に、エンリッチメント解析をやってみる。STRING appには、GO Process / Component / Function、InterPro、KEGG Pathways、PFAMが含まれており、これらの解析が可能らしい。
> string.cmd = 'string retrieve enrichment allNetSpecies="Homo sapiens",
background=genome selectedNodesOnly="false"'
> commandsRun(string.cmd)
> string.cmd = 'string show enrichment'
> commandsRun(string.cmd)
> string.cmd = 'string show charts'
> commandsRun(string.cmd)
・でいけると思ったのだけれど、エラーでうまくいかない。。RCy3はCytoscapeを動かすパッケージなので、Cytoscapeから直に実行してみると、以下のような感じ。
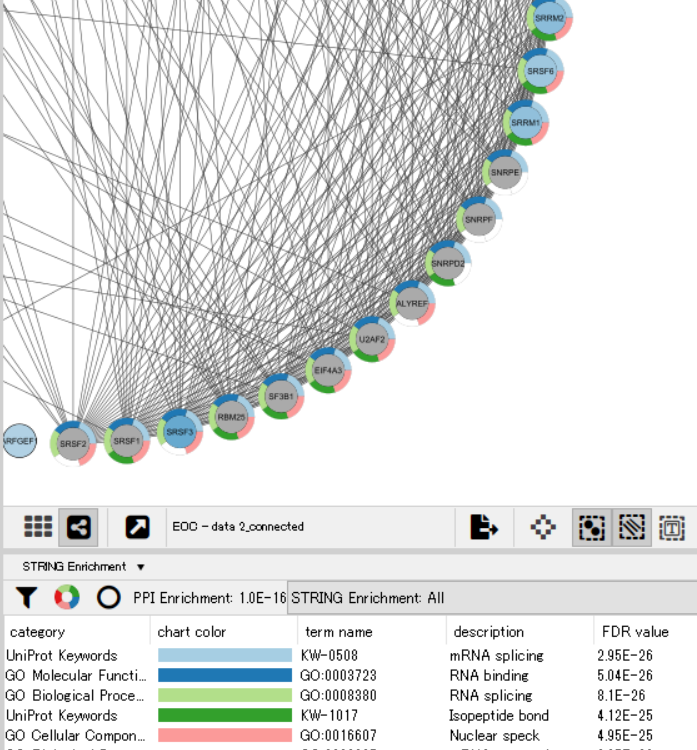
・以下のコマンドで表として保存できるらしい。
> commandsPOST(paste0('table export table="STRING Enrichment: All" options="CSV" outputFile="',paste(getwd(),"string-enrichment-all.csv",sep = "/"),'"'))
おわり。