・先日の続きで、プレートリーダーのデータを解析して、IC50(50%反応阻害濃度)などを求めてみたい。
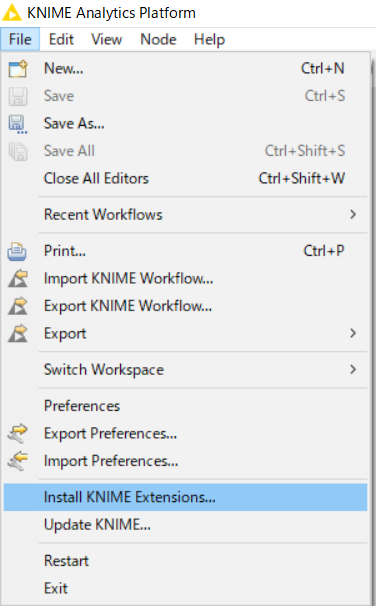
・デフォルトのKNIMEの機能では目的の解析が難しいので、ExtensionのHTS Toolsをインストールする。Extensionは下図のFile⇒install KNIME Extensions...から検索すると辿り着く。
・このExtensionには、Dose Response(R)というノードが存在するのだが、このノードは単独で使えない。まず、Rという統計解析ソフトをインストールする必要がある。
・Rは、以下からダウンロードできる。Rの日本語の説明は、統計局のHPが分かりやすい。
・更に、Rに解析パッケージをインストールする必要がある。
・まず、Rを起動し、CRANミラーサイトを設定する(下図)。
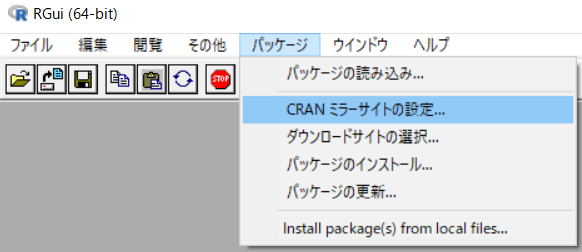
・次に、以下のコマンドで、drcパッケージとRserveパッケージをインストールする。
install.packages("drc")
install.packages("Rserve")
・更に、Rを起動した状態で、Rで以下のコマンドを実行する。
library(Rserve); Rserve(args = "--vanilla")
・これでようやくDose Response(R)ノードを使用できるようになる。
・以前整理した表をそのまま使って、以下のように解析した。
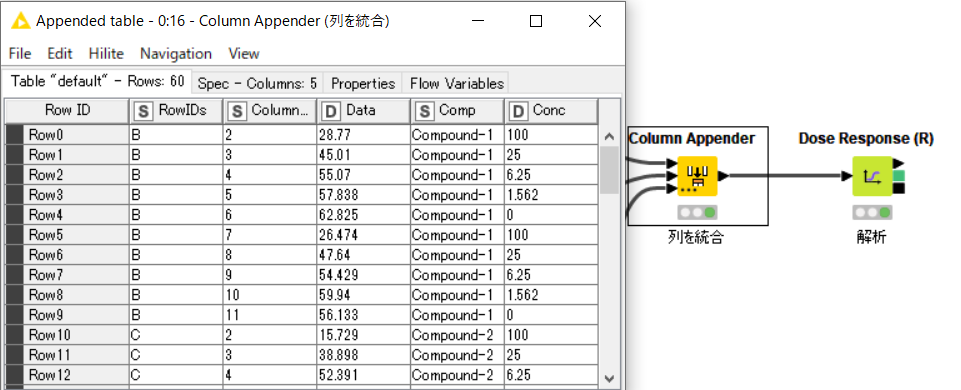
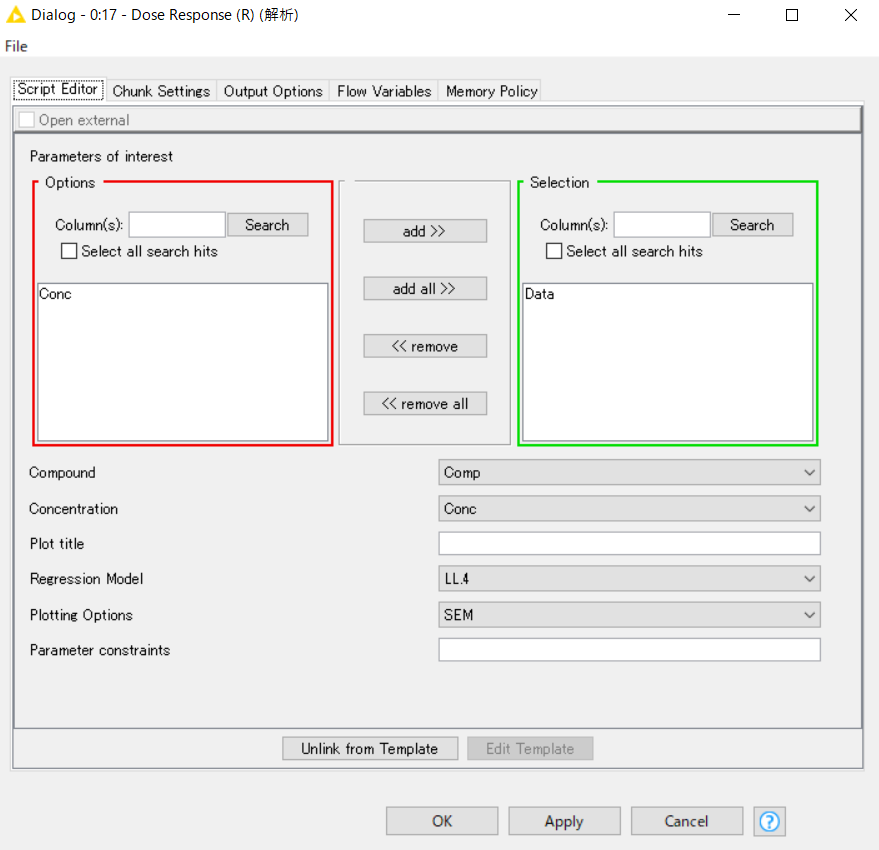
・Dose Response(R)ノードの設定は以下の通り。
・出力の上のポートからは、数値の基本情報、真ん中のポートからはグラフ、下のポートからはモデル情報が出力される(下図)。
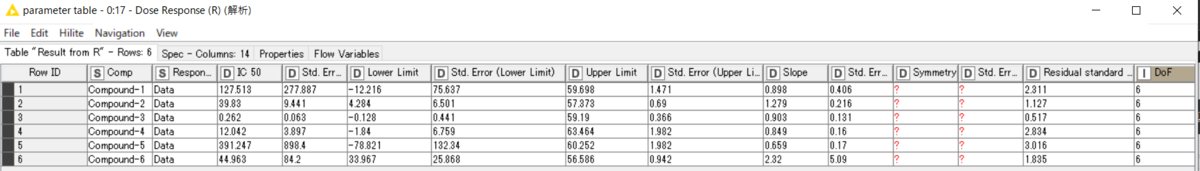
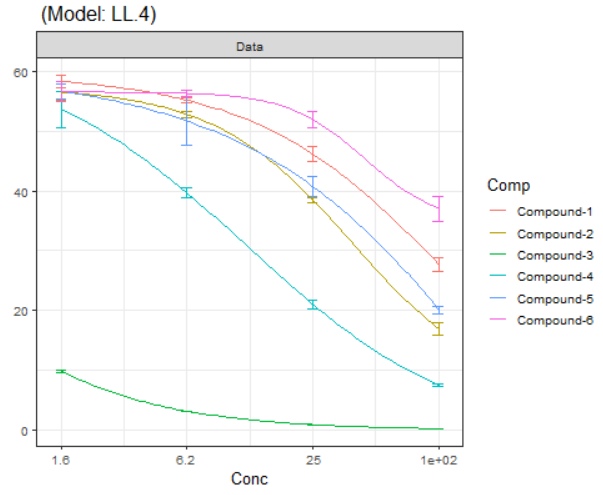
・それっぽく濃度反応性を評価することができたように思う。
・今回はお試し版として、簡易的な実験を設定してみた。細胞の反応を阻害することを想定した実験であれば、100%阻害(細胞なしや刺激誘導なし。今回は設定していない)と0%阻害(試験物質の媒体対照など。今回でいう濃度ゼロ)に相当する条件を設定し、可能であればn=3以上にすることが望ましいと思う。今回は各試験物質に対して0%阻害を設定する設計にしたが、普通ここまでクドく置く必要はないと思う。また、今回は細胞の反応に対する阻害を評価したいので、先に算出した100%阻害と0%阻害のデータから、いったん阻害率を算出してから50%阻害濃度を算出することが望ましいと思う(今回の縦軸は%ではなく、直接的な観測値。今回のシグモイドカーブのトップが60弱なので、30弱になる濃度が50%阻害濃度=IC50に相当する)。更に、0%から100%までの反応が想定できるのならば、今回設定した4パラメーターロジスティック回帰ではなく、2パラメーターロジスティック回帰などの方がそれらしい結果が出るかもしれない。設定方法を調べる必要はあるが。。
・必要に応じて、反応率または阻害率を算出するワークフローと組み合わせて使うと有用と感じた。
終わり。