・お題:生体内の様々な分子は相互作用し、絶妙なバランスを保っている。生体内の分子の役割を理解するのに、ネットワーク解析が有効らしい。学んでみたい。
・私は素人なので、参考サイトをなぞるような形で学んでいきたい。今回は以下のサイトを参考に学ばせていただく。九州工大の竹本先生のHP。日本語でここまで詳しく解説されているHPは他に見たことがないので、ぜひこちらのHPをご覧いただきたい。
・竹本先生の貴著「生物ネットワーク解析」も名著だと思うので、気になる方はぜひチェックしてみてほしい。
・まず、今回使用するパッケージ(igraphとlinkcomm、RColorBrewer)をインストールする(割愛)。
・igraphをアクチベートする。
>library(igraph)
・試しに、シンプルなエッジリストをtextで作成し、カレントディレクトリに保存する。

・これを、グラフとして読み込む。
>g <- read.graph("20230321_igraph.txt", directed = F)
・描画してみる。
>plot(g)

・もちろん、textファイル以外のファイル形式も読み込むことができる。read.graph関数の引数formatにファイル形式("gml"など)を与えれば良いらしい。
・次に、ノード名が文字で与えられている場合を考える。1列目が根元のノード、2列目が行先のノード、3列目が重みで以下のようなtextファイルを作成した。

・この場合、ファイルを表として読み込んだあと、グラフオブジェクトを作成する際に、どの列がノードなのか与えてやる。
> d <- read.table("20230321_igraph.txt")
> g <- graph.data.frame(d[1:2],directed=F)
> plot(g)

・ちなみに、graph.data.frame関数の引数directedにFALSEを与えると無向グラフ、TRUEを与えると有効グラフになる。引数verticesにノードリストを与えれば、エッジを持たないノードも加えられるみたい。
>g <- graph.data.frame(d[1:2],directed=T)
>plot(g)

・グラフオブジェクトがどのように格納されているかというと、以下のような感じ。
> g
IGRAPH 08cb8f9 DNW- 9 7 --
+ attr: name (v/c), weight (e/n)
+ edges from 08cb8f9 (vertex names):
[1] A->B B->C D->C E->F E->G E->H E->I
・ノードはV()で、エッジはE()で確認することができる。
> V(g)
+ 9/9 vertices, named, from 08cb8f9:
[1] A B D E C F G H I
> E(g)
+ 7/7 edges from 08cb8f9 (vertex names):
[1] A->B B->C D->C E->F E->G E->H E->I
・エッジに重みを与えるには、E()のweightに重みを与えてやれば良い。
>E(g)$weight <- d[ [3] ]
・ノード名はV()のnameに格納されている。
> V(g)$name
[1] "A" "B" "D" "E" "C" "F" "G" "H" "I"
・次に、以下のようなマトリックスでデータを与えてみる。

・ファイルをマトリックスとして読み込む。
>d <- as.matrix(read.table("20230331_igraph2.txt",header=T,row.names=1))

・重み付き有効グラフとしてグラフオブジェクトに読み込む。無向グラフ、重みなしグラフの場合、引数modeに"undirected"、引数weightedにNULLを与えれば良い。
>g <- graph.adjacency(d, mode="directed", weighted=T)
>plot(g)

・自己ループやループの重複を除去することもできる。
>g <- simplify(g,remove.multiple=T,remove.loops=T)
>plot(g)

・ちなみに、plot関数の引数にいろいろ指定すると、グラフの描画をもっときれいにできるらしい。代表的なもので、vertex.size(ノードの大きさ)、vertex.shape(ノードの形)、vertex.label(ノードのラベル)、vertex.color(ノードの色)、vertex.label.cex(ノードラベルの文字サイズ)、edge.width(エッジの太さ)、edge.color(エッジの色)、layout(レイアウト)などがあるらしい(一部割愛)。
・ここまでで、ネットワークを読み込んでグラフを描けるようになったわけだけれど、本当にやりたいのは、ネットワークから意味のある情報を抽出することだと思う。ということで、大事なノードを探してみたい。大事さ、をネットワーク業界では中心性と呼ぶらしい。中心性にはいろいろな種類があるので、いろいろ試してみたい。
・例として、以下のようなサンプルデータを作成し、textファイルでカレントディレクトリに保存した。
A C 1
D C 2
C B 3
E C 2
C G 3
C H 4
G F 3
G I 1
H I 2
I Q 1
Q R 2
R M 3
R K 4
J K 4
K L 3
K M 3
L M 2
M O 2
M P 3
・とりあえず、そのままで描画する。
> d0 <- read.table("20230321_igraph3.txt")
> g0 <- graph.data.frame(d0[1:2],directed=T)
> E(g0)$weight <- d0[ [3] ]
> set.seed(1)
> layout <- layout.fruchterman.reingold(g0)
> plot(g0, layout=layout)

・各ノードの次数(何本エッジが伸びているか)を見てみる。
> degree(g0)
A D C E G H I Q R J K L M B F O P
1 1 6 1 3 2 3 2 3 1 4 2 5 1 1 1 1
・これを使うと、次数中心性というのが出せる。
> dc <- degree(g0) / (vcount(g0) - 1)
> dc

・これをもとに、ネットワークグラフを描いてみる。
> class <- cut(dc,breaks=9)
> clscolor <- data.frame(class=levels(as.factor(class)),
+ color=I(brewer.pal(nlevels(as.factor(class)),"OrRd")))
> plot(g0,
+ vertex.color=clscolor$color[match(class,clscolor$class)],
+ layout=layout)
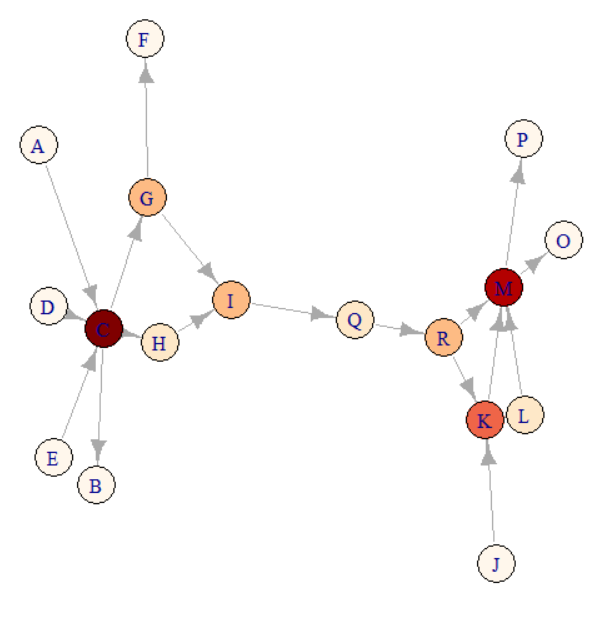
・媒介中心性
> bc <- betweenness(g0)
> bc
A D C E G H I Q R J K L M B F O P
0 0 36 0 36 0 42 42 40 0 13 0 24 0 0 0 0
> class <- cut(bc,breaks=9)
> clscolor <- data.frame(class=levels(as.factor(class)),
+ color=I(brewer.pal(nlevels(as.factor(class)),"OrRd")))
> plot(g0,
+ vertex.color=clscolor$color[match(class,clscolor$class)],
+ layout=layout)

・近接中心性
> cc <- closeness(g0)
> cc

> class <- cut(cc,breaks=9)
> clscolor <- data.frame(class=levels(as.factor(class)),
+ color=I(brewer.pal(nlevels(as.factor(class)),"OrRd")))
> plot(g0,
+ vertex.color=clscolor$color[match(class,clscolor$class)],
+ layout=layout)

・固有ベクトル中心性
> ec <- evcent(g0)$vector
> ec

> class <- cut(ec,breaks=9)
> clscolor <- data.frame(class=levels(as.factor(class)),
+ color=I(brewer.pal(nlevels(as.factor(class)),"OrRd")))
> plot(g0,
+ vertex.color=clscolor$color[match(class,clscolor$class)],
+ layout=layout)

・Page Rank(の一種)
> pr <- page.rank(g0)$vector
> pr

> class <- cut(pr,breaks=9)
> clscolor <- data.frame(class=levels(as.factor(class)),
+ color=I(brewer.pal(nlevels(as.factor(class)),"OrRd")))
> plot(g0,
+ vertex.color=clscolor$color[match(class,clscolor$class)],
+ layout=layout)

・それぞれの中心性の意味に関しては、以下のサイトがイメージしやすかったように思うので、ご参照いただければと思う。
・個人的には、媒介中心性とRank Pageが気になる今日この頃。
つづく。